1)首先在一特定的空间进行空间剖分获得每一个晶核坐标对应的随机数,借助MATLAB里面的Multi-Parametric Toolbox(MPT)工具箱赋有的 mpt_voronoi函数命令,生成带有拓扑信息且具有指定大小和晶粒数目的三维Voronoi多晶体示意图。
2)在ABAQUS/CAE中建立一个与MATLAB构建的三维Voronoi多晶体示意图相同大小的Part模型,并划上适量数目的网格单元,根据每一个单元编号的空间排布特点尤其是单元编号的排列规律,直接依次求得每一个单元体形心坐标。
3)求得每一个单元体的形心坐标后,接下来就是依次求取每一个单元的形心坐标与每个晶核的距离,最终把每一个单元归属于其形心坐标距离最近晶核所处的晶粒中。
4)把上述所得的每一个晶粒的编号和该晶粒包含的全部单元编号,最后用多级列表的方式存放于txt文本里,为后面修改INP文件做好准备工作。
经过上面MATLAB部分的编程,仅仅只是得到了关于三维Voronoi图的全部拓扑结构信息。为此必须得在ABAQUS生成的INP文件里的part部分编写与晶粒数相同多的set集合,每一个set集合就作为一个晶粒。接着,在txt文本里找到该晶粒包含的所有单元编号写入与之对应的set集合中完成整个建模。在INP文件里实现三维建模的具体步骤如下:
1)于ABAQUS/CAE模块中创建单个3D/Deformable/Shell实体,选择默认装配,划分网格类型为C3D8R的网格单元,接着在Job模块中点击Write Input命令生成相应的INP文件。
2)把之前所得的每一个晶粒的编号和该晶粒包含的全部单元编号作为一个set集合,最终把每一个set集合以多级列表的方式写入已建立好的INP文件里对应的part部分。在ABAQUS里重新打开修改之后的模型(INP文件格式类型),并且在Color Code Dialog中选择Color code by:Sets。
这里基本介绍了如何在MATLAB中建立三维voronoi后再导入到二维中的详细过程,但是总感觉这个过程比较繁琐,另外也有通过python二次开发进行直接在ABAQUS中建立模型,具有代表性的的就是星辰北极星团队开发的voronoi插件,使用起来非常方便,当然,个人也做了一个生成voronoi3d晶粒的程序,但是,这些都是先建立几何模型后再进行网格剖分,网格的边界和晶粒边界一致,这样对于三维voronoi来说,由于结构比较复杂,网格划分起来往往都比较困难,要不然就是单元少网格不好,要不然就是网格还可以但单元太多了,这就比较纠结了,所以,我们想着通过现在也是比较流行的映射网格来划分voronoi体,这样就不会出现上面单元数量与单元质量之间的矛盾了,这方面具有代表性的是neper软件,但是neper是一个基于linux的小众软件,大部分人都不懂或赖得使用,这里我们介绍一款ABAQUS的voronoi映射网格生成插件,二维的其实比较好做,只要生成了二维voronoi图片,然后,结合我们前期帖子中介绍的基于实际形貌的映射网格生成方法即可得到,但是对于三维的模型,因为是3d空间,这种方法就无能为力了,因此只能通过二次开发编程实现。
二维模型:
插件程序及源码图示:
macroVoronoi.pyc
voronoi.pyc
voronoiDB.pyc
voronoiIcon.pyc
Voronoy_plugin.pyc
*
macroVoronoi.py
voronoi.py
voronoiDB.py
voronoiIcon.py
Voronoy_plugin.py
生成的几何模型:
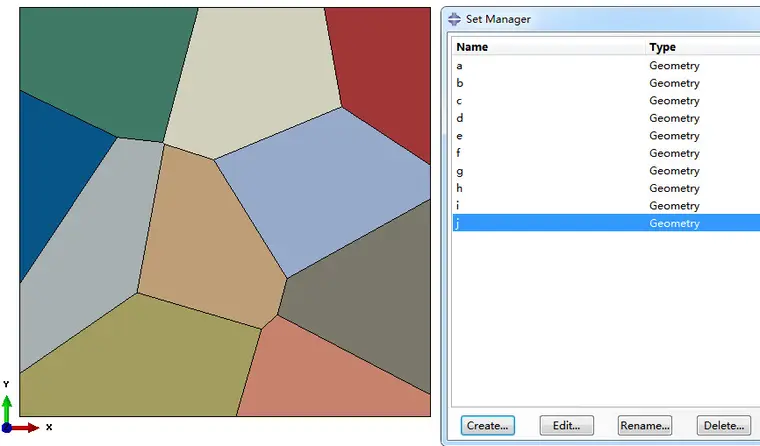
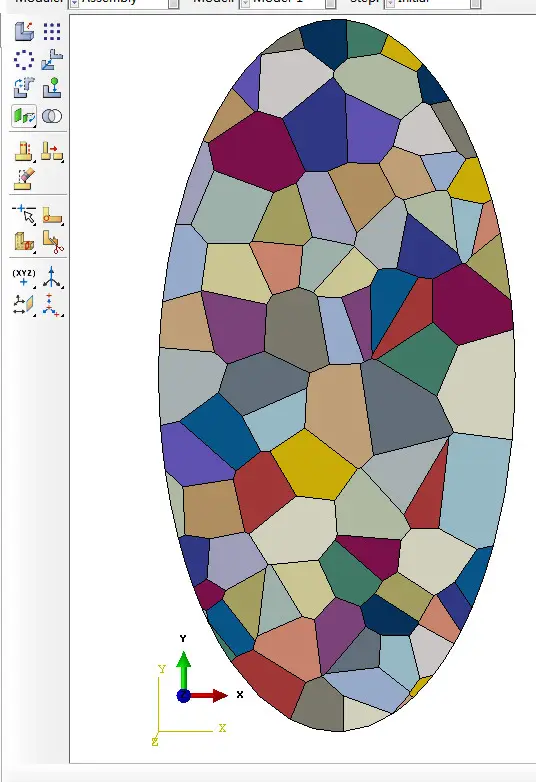
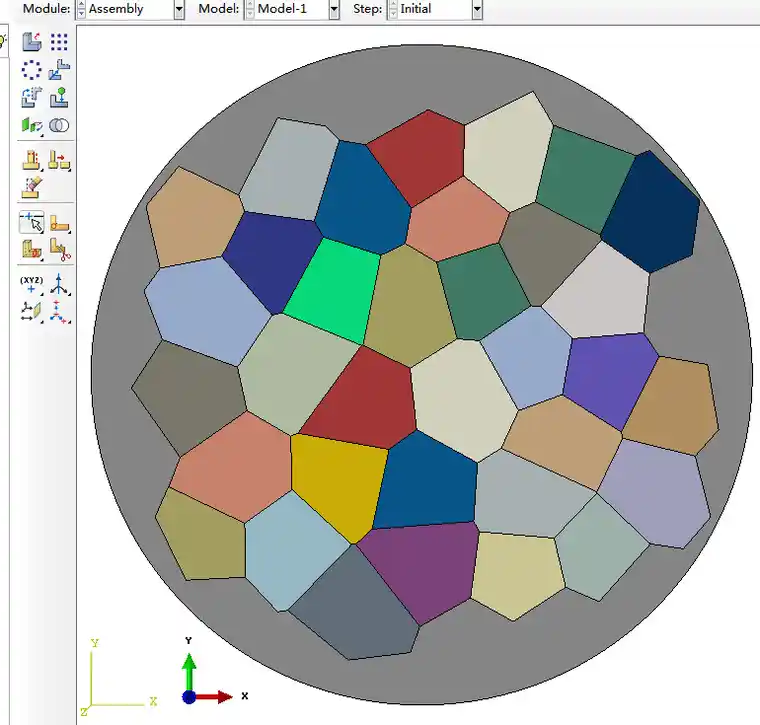
生成的映射网格(不区分晶界区域):
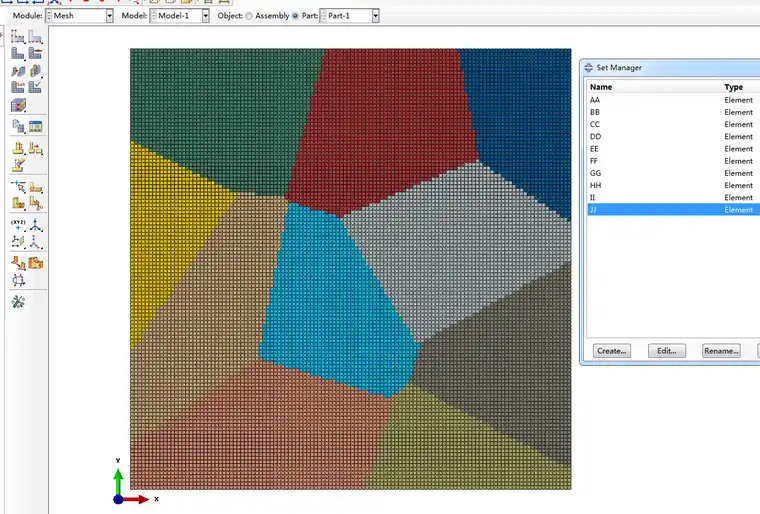
生成的映射网格(区分晶界区域):
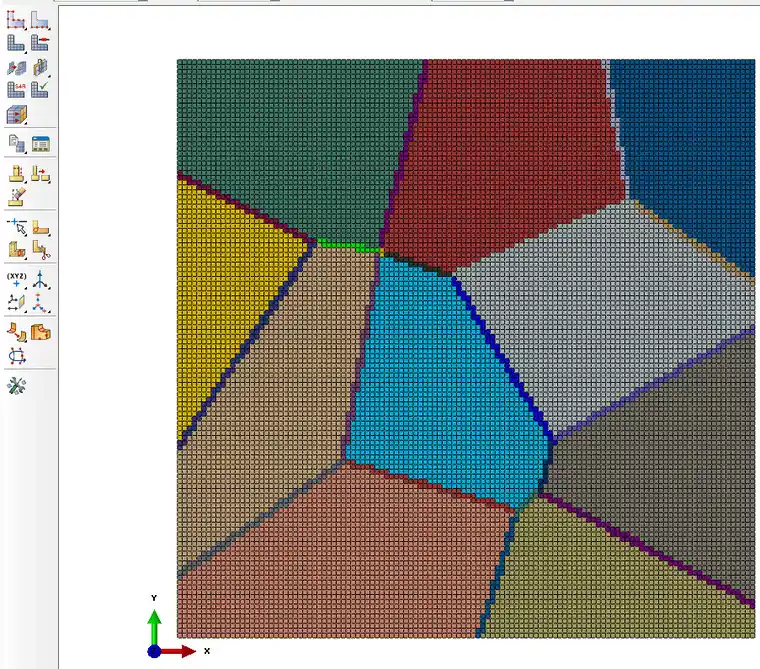
三维模型:
插件程序及源码图示:

#n为种子点,晶粒数、
r1,r2,r3为长、宽、高
#mo为杨氏模量,po为泊松比
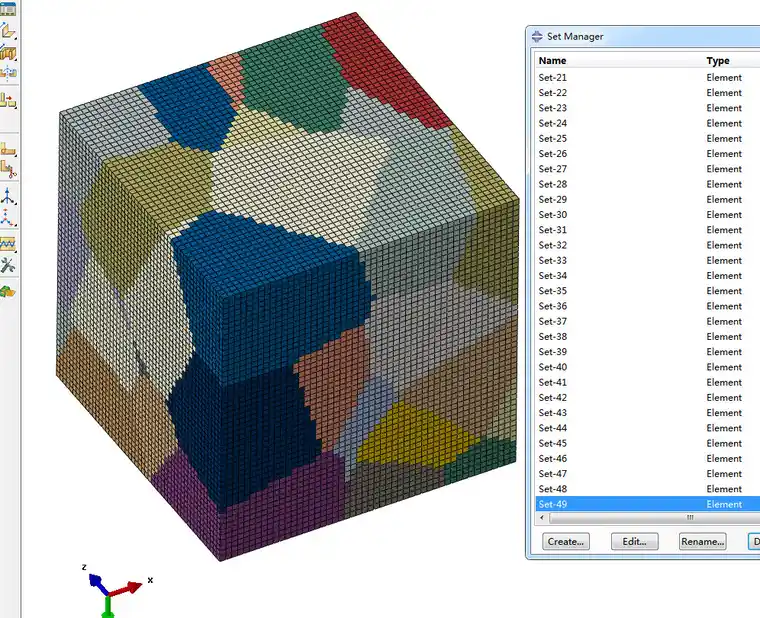
生成的映射网格(不区分晶界区域):
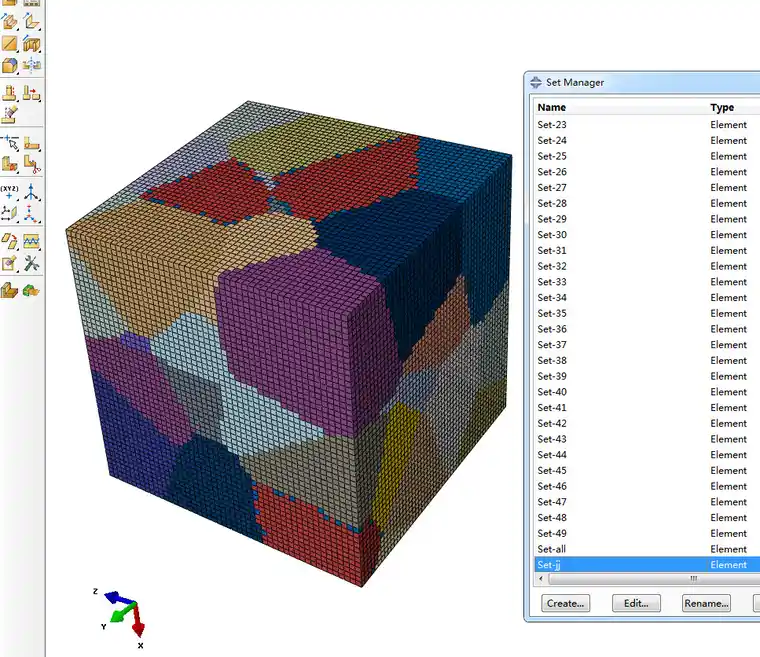
生成的映射网格(取边界上的1/3单元作为增强颗粒):
使用插件可以极快速分分钟生成想要的voronoi模型,而且这种网格划分不会造成计算困难,对后续的分析提供了便利。
免责声明:本文系网络转载或改编,未找到原创作者,版权归原作者所有。如涉及版权,请联系删