实验室新的服务器组开始使用Scientific Linux release 7.9 (Nitrogen)作为操作系统,传说中Abaqus求解器在Linux下资源利用效率更高,不过对我们这坨从小学开始学微软的老用户就有些不友好了……幸好带GUI的Linux用起来与Windows差异不大,但之前在Windows下用来批量提交的那坨脚本显然就没法用了,不得已开始用Bash写新的批处理,好消息是代码改动并不多。
在Linux似乎拖放提交这种操作是不简单了,考虑下来主要采用两种方法进行批量提交,同样还是写文本文档将下方代码写入后保存sh格式文件。

方案一:终端调用该sh脚本来进行批量提交
#!/bin/bash
export PATH=/var/DassaultSystemes/SIMULIA/Commands/:$PATH
pushd $1
for file in `ls $1/*.inp`;
do
abaqus job=`basename $file` cpus=6 gpus=1 mp_mode=thread int ask_delete=OFF;
done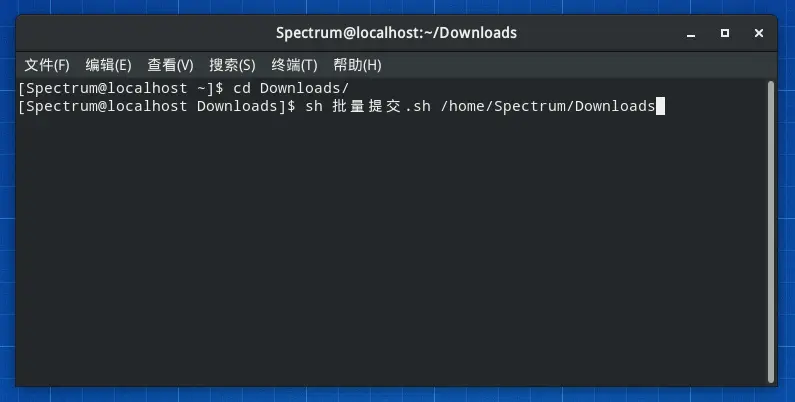

方案二:双击sh脚本提交该脚本所在文件夹中的所有INP文件
#!/bin/bash
export PATH=/var/DassaultSystemes/SIMULIA/Commands/:$PATH
pasway=`dirname $0`
pushd $pasway
for file in `ls $pasway/*.inp`;
do
abaqus job=`basename $file` cpus=6 gpus=1 mp_mode=thread int ask_delete=OFF;
done
echo 计算结束,按任意键退出
read -n 1

补充:双击sh脚本提交该脚本所在目录下所有子文件夹中的INP文件
#!/bin/bash
export PATH=/var/DassaultSystemes/SIMULIA/Commands/:$PATH
pasway=`dirname $0`
cd $pasway
for dire in `ls -d */`;
do
cd $dire
for file in `ls *.inp`;
do
abaqus job=`basename $file` cpus=6 gpus=1 mp_mode=thread int ask_delete=OFF;
done
cd ..
done
echo 计算结束,按任意键退出
read -n 1也可以通过简单修改将这个代码匹配联合仿真:
#!/bin/bash
export PATH=/var/DassaultSystemes/SIMULIA/Commands/:$PATH
pasway=`dirname $0`
cd $pasway
for dire in `ls -d */`;
do
cd $dire
abaqus cosimulation cosimjob=Co-execution-1 job=Co-execution-1-Project_01,Co-execution-1-CSF-2 configure=Co-execution-1_config.xml cpus=6,6 double=both,off int ask_delete=OFF;
cd ..
done
echo 计算结束,按任意键退出
read -n 1补充信息:
参考资料同前两次文章。
武汉格发信息技术有限公司,格发许可优化管理系统可以帮你评估贵公司软件许可的真实需求,再低成本合规性管理软件许可,帮助贵司提高软件投资回报率,为软件采购、使用提供科学决策依据。支持的软件有: CAD,CAE,PDM,PLM,Catia,Ugnx, AutoCAD, Pro/E, Solidworks ,Hyperworks, Protel,CAXA,OpenWorks LandMark,MATLAB,Enovia,Winchill,TeamCenter,MathCAD,Ansys, Abaqus,ls-dyna, Fluent, MSC,Bentley,License,UG,ug,catia,Dassault Systèmes,AutoDesk,Altair,autocad,PTC,SolidWorks,Ansys,Siemens PLM Software,Paradigm,Mathworks,Borland,AVEVA,ESRI,hP,Solibri,Progman,Leica,Cadence,IBM,SIMULIA,Citrix,Sybase,Schlumberger,MSC Products...