异常定义为偏离标准,很少发生且不遵循其余“模式”的事件。异常的例子包括:
假设我们的数据服从一个正太分布,那么通常异常数据位于正态分布曲线的两侧。如下图所示。
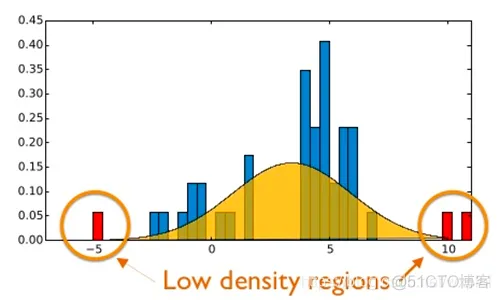
正如我们看到的那样,这些事件将发生,但发生的可能性极低。从机器学习的角度来看,这使得很难检测异常-根据定义,我们有很多“标准”事件的示例,而很少有“异常”事件的示例。因此,我们的数据集存在很大的偏差。当我们要检测的异常可能仅在1%,0.1%或0.0001%的时间发生时,应该如何在平衡数据集中最佳地工作的机器学习算法如何工作?这时候我们就需要用到异常检测来专门处理这类问题。
由于我们的数据集标签存在极大的不平衡,根据定义异常数据很少发生,而我们拥有大量的正常数据。而为了检测异常数据,传统的机器学习算法衍生出如孤立森林、One-class SVMs、Elliptic Envelopes和局部异常因子算法等。这里不一一介绍,有兴趣的同学可以去研究研究,这里主要讲的是如何使用深度学习来解决这个问题。
自动编码器是一种无监督的神经网络,它可以:
通常自动编码器主要有两部分组成:编码器和解码器。编码器接受输入数据并将它转化为特征表示。然后,解码器尝试对压缩的特征进行重构得到输入数据。当我们以端到端的方式训练自动编码器时,该网络能够学习到一种强大的过滤器,其甚至能够对输入数据去噪。
从异常检测角度来看,自动编码器特别之处在于其能够重构损失,在训练自动编码器时通常会衡量输入和重构数据之间的均方误差(MSE)。如果损失越小,那么重构的图像越像原始数据。

假设我们在整个MNIST数据集上训练了一个自动编码器,然后,对自动编码器提供一个数字,并对他进行重构。我们希望我们能够对其进行重建与输入数据相似,然后我们会发现这两张图像之间的MSE比较低。

如果,此时我们再提供一个非数字的图像,如大象,此时这两张图片的MSE非常高。这是由于自动编码器以前从未见过大象,更重要的是,从未接受过重建大象的训练,因此 我们的MSE 很高。
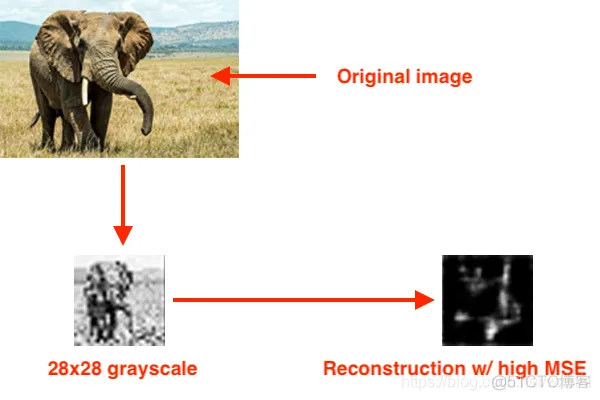
此时,如果重建的MSE高,那么我们可能会有一个异常值。
这里我们使用mnist数据集,将标签为1的作为正常数据,将标签为数据的数量1%的数量的且标签为3的数据作为异常数据,并制作异常数据。具体如下:
# 加载MNIST数据集
print("[INFO] loading MNIST dataset...")
((trainX,trainY),(testX,testY))=tf.keras.datasets.mnist.load_data()
def build_unsupervised_dataset(data,labels,validLabel=1,
anomalyLabel=3, contam=0.01, seed=42):
'''制作数据集'''
# 抓取提供的类标签的所有 * 真正 * 索引特定的标签,
# 然后获取图像的索引个标签将成为我们的“异常”
validIdxs=np.where(labels==validLabel)[0]
anomalyLabelIdx=np.where(labels==anomalyLabel)[0]
#随机打乱数据
random.shuffle(validIdxs)
random.shuffle(anomalyLabelIdx)
#计算并设置异常数据的个数
i=int(len(validIdxs)*contam)
anomalyLabelIdx=anomalyLabelIdx[:i]
#提取正常数据和异常数据
validImages=data[validIdxs]
anomalyImages=data[anomalyLabelIdx]
#打包数据并进行数据打乱
images=np.vstack([validImages,anomalyImages])
return images
# 建立少量的无监督图像数据集,污染(即异常)添加到其中
print("[INFO] creating unsupervised dataset...")
images = build_unsupervised_dataset(trainX, trainY, validLabel=1,
anomalyLabel=3, contam=0.01)
这里直接上代码:
class ConvAutoencoder:
@staticmethod
def build(width,height,depth=None,filters=(32,64),latentDim=16):
'''
构建自动编码器模型
:param width: 图像的宽度
:param height: 图像的高度
:param depth: 图像的深度
:param filters: 卷积核的尺寸
:param latentDim: 全连接的维数
:return:
'''
#定义解码器
inputs=tf.keras.layers.Input(shape=(height,width,depth))
x=inputs
for filter in filters:
x=tf.keras.layers.Conv2D(filter,kernel_size=(3,3),strides=2,padding='same')(x)
x=tf.keras.layers.LeakyReLU(alpha=0.2)(x)
x=tf.keras.layers.BatchNormalization(axis=-1)(x)
volumeSize=tf.keras.backend.int_shape(x)
x=tf.keras.layers.Flatten()(x)
latent=tf.keras.layers.Dense(latentDim)(x)
encoder=tf.keras.Model(inputs=inputs,outputs=latent,name='encoder')
#定义编码器
latentinputs=tf.keras.layers.Input(shape=(latentDim,))
x=tf.keras.layers.Dense(np.prod(volumeSize[1:]))(latentinputs)
x=tf.keras.layers.Reshape((volumeSize[1],volumeSize[2],volumeSize[3]))(x)
for filter in filters[::-1]:
x=tf.keras.layers.Conv2DTranspose(filter,kernel_size=(3,3),strides=2,padding='same')(x)
x=tf.keras.layers.LeakyReLU(alpha=0.2)(x)
x=tf.keras.layers.BatchNormalization(axis=-1)(x)
x=tf.keras.layers.Conv2DTranspose(depth,(3,3),padding='same')(x)
outputs=tf.keras.layers.Activation('sigmoid')(x)
decoder=tf.keras.Model(latentinputs,outputs,name='decoder')
autoencoder=tf.keras.Model(inputs,decoder(encoder(inputs)),name='autoencoder')
return (encoder,decoder,autoencoder)
autoencoder.summary()
网络框架结构如下:

epochs=20
lr=1e-3
batch_size=32
#搭建模型
print("[INFO] building autoencoder...")
(encoder, decoder, autoencoder) = ConvAutoencoder.build(28, 28,1)
#搭建优化器
opt=tf.keras.optimizers.Adam(lr=lr,decay=lr/epochs)
autoencoder.compile(loss='mse',optimizer=opt,metrics=['acc'])
#训练
H=autoencoder.fit(trainX,trainX,validation_data=(testX,testX),epochs=epochs,batch_size=batch_size)
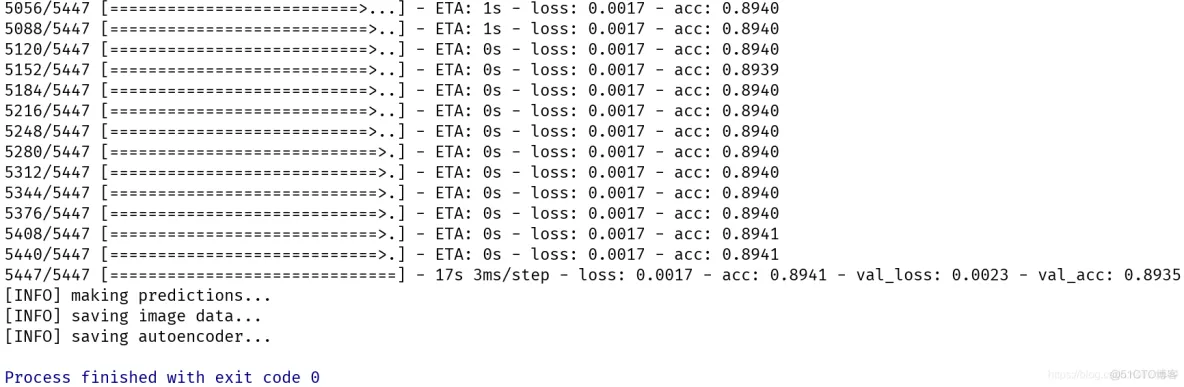
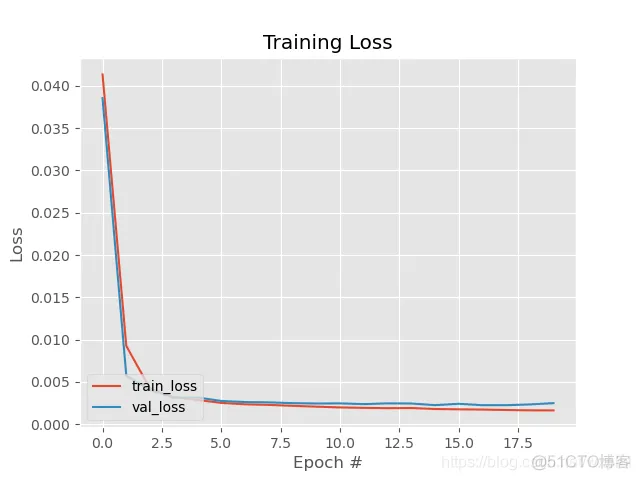
训练过程如下:
主要使用自编码重构后的图片与原图片计算MSE,然后根据阈值进行相应的判断,主要实现如下:
# 加载模型
print("[INFO] loading autoencoder and image data...")
autoencoder = tf.keras.models.load_model("autoencoder.h5")
images = pickle.loads(open("mages.pickle", "rb").read())
#预测图片
images=images.reshape(-1,28,28,1)
images=images.astype('float32')
images/=255
decoded = autoencoder.predict(images)
errors = []
for (image, recon) in zip(images, decoded):
# 计算预测和真实图片之间的均方差1
mse = np.mean((image - recon) ** 2)
errors.append(mse)
# compute the q-th quantile of the errors which serves as our
# threshold to identify anomalies -- any data point that our model
# reconstructed with > threshold error will be marked as an outlier
thresh = np.quantile(errors, 0.999)
idxs = np.where(np.array(errors) >= thresh)[0]
print("[INFO] mse threshold: {}".format(thresh))
print("[INFO] {} outliers found".format(len(idxs)))
测试结果如下:

免责声明:本文系网络转载或改编,未找到原创作者,版权归原作者所有。如涉及版权,请联系删