病人层次的”CMAP”——首个病人来源的癌症药物扰动基因表达信号数据库CDS-DB,助力癌症药物重定向、联合用药发现
CMAP[1]和LINCS L1000[2]是众所周知的细胞系层次的药物分子扰动信号资源,通过信号连通性分析,该资源已经被广泛应用于药物重定向、靶标发现等。然而,细胞系和病人样本存在显著差距。目前尚无病人来源的癌症药物引起的基因表达信号资源。
CDS-DB是国家蛋白质科学中心(北京)发展的首个病人来源的癌症药物引起的基因表达信号数据资源,成果发表在Nucleic Acids Res上。数据库收录了来自GEO/ArrayExpress中78个配对的用药前、后的临床转录组源数据集,后将源数据集划分为181个数据集级别的基因扰动信号,2012个患者级别的基因扰动信号。这些数据共涉及85个治疗方案、71种药物、39种癌症亚型以及3628个病人样本。其中57%的数据集级别扰动信号和45%的病人级别扰动信号有药物响应信息。为了更好地利用上述数据,除了数据浏览、检索和下载,数据库还支持单信号分析、多信号比较分析、信号连通性分析等功能。这些数据以及丰富的数据分析功能有助于揭示癌症患者药物治疗的作用机制,及其在患者中的同质性/异质性、药物(联合)发现等。
注解:CDS-DB数据集:将从GEO/ArrayExpress手工挖掘得到的成对的用药前后临床转录组源数据集(source dataset)进一步划分为CDS-DB数据集。每个CDS-DB数据集由配对治疗前后病人样本组成,这些样本使用相同的治疗方案和剂量,具有相同癌症亚型、相同的取样部位分类(组织/血液/骨髓/腹水)、相似的治疗后取样时间和相同的响应分组(响应/不响应)(如果此信息可用)。
CDS-DB数据集级别基因扰动信号:获得自CDS-DB数据集中成对的用药前、后临床转录组数据的差异基因表达分析。
CDS-DB患者级别基因扰动信号:获得自一个病人的一对用药前、后转录组数据的差异基因表达分析。
在这篇博文中,我们重点介绍一下信号连通性分析功能。其他分析功能介绍详见首个病人来源的癌症药物扰动基因表达信号数据库CDS-DB,助力揭示癌症药物在患者体内的MOA。通过CDS-DB数据库的"Analyze"功能,用户可以访问信号连通性分析工作流。利用该工作流,用户可以实现基于CDS-DB信号的(联合)药物发现、药物重定向等。
针对用户提交的信号,该工作流将返回经过排序的能够“模拟”或者“逆转”用户提交信号的CDS-DB信号,进而分析返回的信号的药物作用机制(MOA)和靶标偏好。根据用户提交的信号,该工作流程适用于多种生物医学场景。例如,当用户提交(源自患者的)疾病信号时,可以通过搜索能够逆转该疾病信号的CDS-DB药物扰动信号,从而发现能够治疗该疾病的潜在药物和靶点/MOA。而当用户提交(源自患者的)药物A的抵抗信号时,可以通过搜索能够逆转其的CDS-DB药物扰动信号来发现能够克服药物A抵抗的增敏药物B。
了解到病人来源的连通性分析的使用场景,接下来我们看看应该如何使用该功能。
“Analyze”界面支持三种输入类型:(i)"基因集",需要提供上调和下调基因列表;(ii)"信号",需要提供带有logFCs的基因列表;(iii)"细胞类型",需要提供上调和下调细胞类型列表。对于前两种输入类型,输入信号与CDS-DB基因扰动信号进行比对,而对于最后一种输入类型,则与细胞扰动信号进行比对。细胞扰动信号通过MCP-counter和xCell两种方法获取,因此用户需要在此选择方法。MCP-counter和xCell分别支持10种和64种细胞类型。此外,用户还可以点击"CTR-DB患者来源的药物抵抗信号"按钮,从CTR-DB中选择患者来源的药物抵抗信号作为输入的"信号"。CTR-DB是该工作团队之前开发的病人来源的具有癌症治疗响应的临床转录组数据库。通过对无响应和有响应的患者的基线(即治疗前)转录组进行差异基因表达分析获得药物的抵抗基因表达信号[3]。在信号连通性分析中,可选参数包括信号水平(CDS-DB数据集或患者水平信号)和方向(模拟或逆转)。
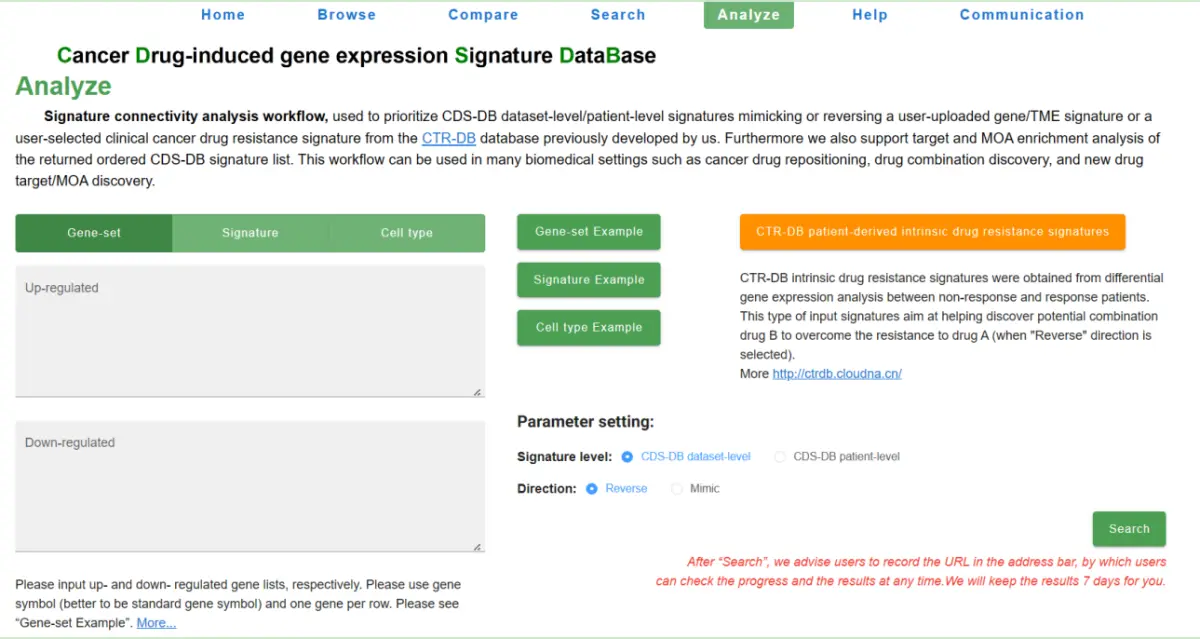
下面举例说明该功能的应用。
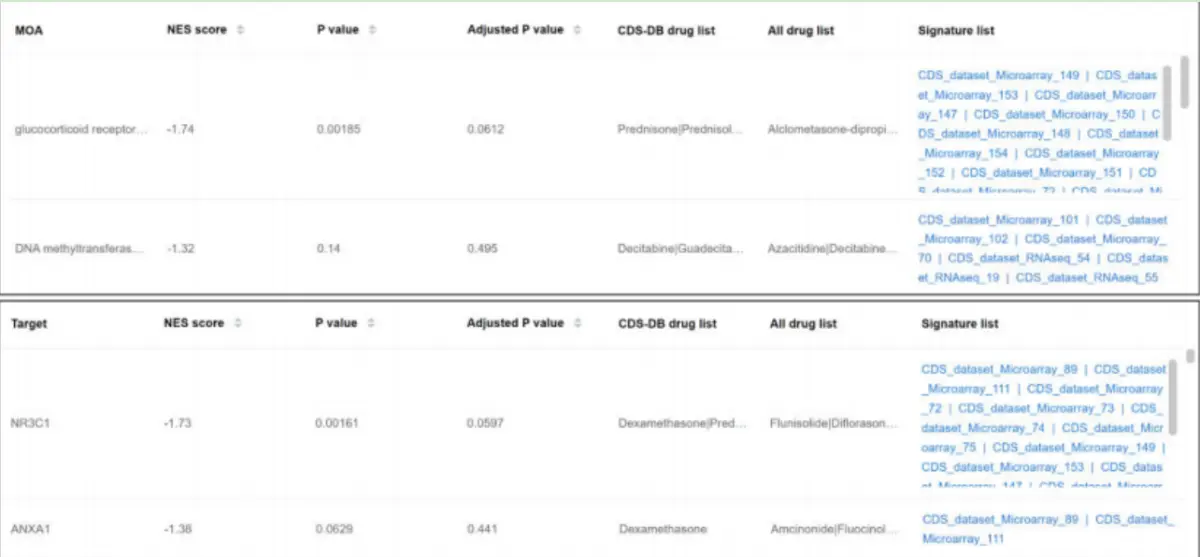
在“Analyze”界面,以急性淋巴细胞白血病(ALL)疾病信号作为输入(获得过程详见CDS-DB文章附录),搜索逆转它的CDS-DB信号后,发现排名靠前的CDS-DB药物扰动信号中显著富集的MOA是糖皮质激素受体激动剂,(NES =−1.74,P-value = 1.85E−03),显著富集的靶标是NR3C1(即糖皮质激素受体)(NES=−1.73,P-value= 1.61E−03)。我们都知道在临床ALL治疗中,糖皮质激素是化疗方案中必不可少的药[4],这表明这一工作流程在癌症药物发现方面的潜力。
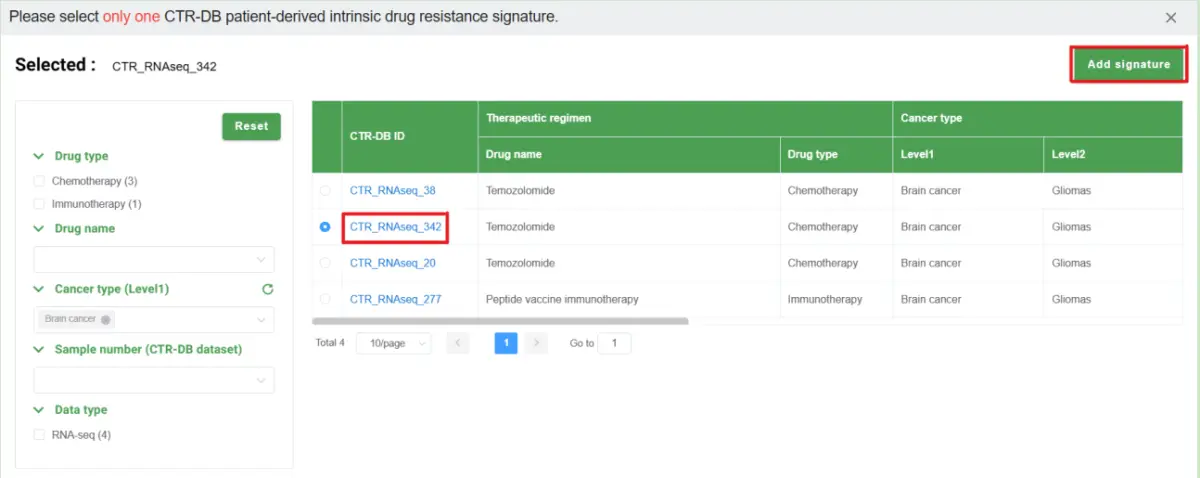
为了进一步证明该工作流程在药物联合发现中的潜力,我们从CTR-DB数据库中选择病人来源的药物抵抗信号CTR_RNAseq_342作为输入。CTR_RNAseq_342获得自接受替莫唑胺治疗的38例少突胶质细胞瘤患者的治疗前转录组数据,通过对其中10例应答者和28例耐药者的差异基因表达分析获得。
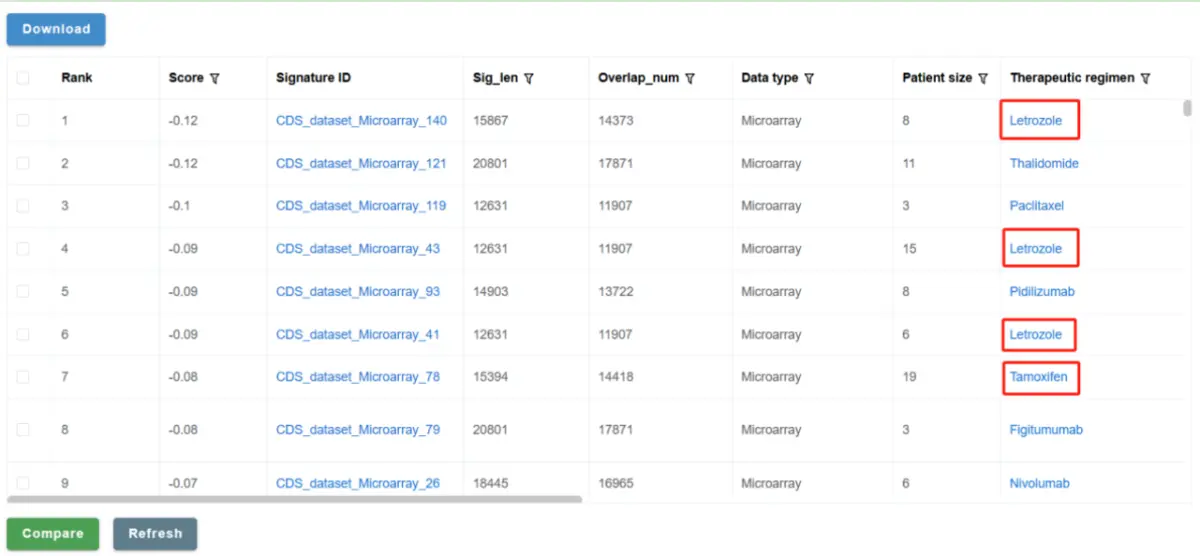
我们使用“Analyze”来寻找能够“逆转”该抵抗信号的CDS-DB信号,以发现能够克服替莫唑胺耐药的潜在联合药物B。结果发现,在数据集级别,返回的前10个信号中有3个与来曲唑相关,其中一个与他莫昔芬相关。来曲唑是一种芳香化酶抑制剂,其中芳香化酶也被称为雌激素合成酶。他莫昔芬是一种雌激素受体拮抗剂。这些结果提示,雌激素和雌激素受体可能是克服替莫唑胺耐药的重要靶点。最近的研究也确实为这一发现提供了证据支持:最近的临床前研究发现来曲唑[5]和他莫昔芬[6]可能在胶质瘤的治疗中发挥重要作用,来曲唑/他莫昔芬和替莫唑胺在胶质母细胞瘤细胞系中具有协同作用[7-8]。已经进行了一项I期临床试验来测试他莫昔芬和替莫唑胺联合治疗胶质瘤[9],Cristofori等人认为他莫昔芬可能会增加替莫唑胺治疗复发性胶质母细胞瘤的疗效[10]。
基于CDS-DB信号的连通性分析能够从一个新的维度(即患者水平)为(联合)药物发现和药物重定向提供线索。这些线索可以验证和补充目前常用的细胞系水平药物扰动信号(如CMAP或LINCS L1000)分析。此外,当用户输入源自患者的信号时,基于同样来自患者样本的CDS-DB药物扰动信号的分析可能会展现更卓越的性能。
CDS-DB数据库除了可以进行患者来源的信号连通性分析,以发现(联合)药物和药物重定向外,还支持单信号分析功能(包括差异基因表达、功能富集、肿瘤微环境和相关性分析),和信号比较分析功能,可以对药物MOA及其在患者中的同质性和异质性进行探索。对此感兴趣的小伙伴们快去试试吧!
参考文献:
[1] Lamb, J., Crawford, E. D., Peck, D., Modell, J. W., Blat, I. C., Wrobel, M. J., Lerner, J., Brunet, J. P., Subramanian, A., Ross, K. N. et al. (2006) The Connectivity Map: using gene-expression signatures to connect small molecules, genes, and disease. Science, 313, 1929–1935.
[2] Subramanian, A., Narayan, R., Corsello, S. M., Peck, D. D., Natoli, T. E., Lu, X., Gould, J., Davis, J. F., Tubelli, A. A., Asiedu, J. K. et al. (2017) A Next Generation Connectivity Map: L1000 Platform and the First 1,000,000 Profiles. Cell, 171, 1437–1452.e17.
[3] Liu, Z., Liu, J., Liu, X., Wang, X., Xie, Q., Zhang, X., Kong, X., He, M., Yang, Y., Deng, X. et al. (2022) CTR-DB, an omnibus for patient-derived gene expression signatures correlated with cancer drug response. Nucleic Acids Res., 50, D1184–D1199.
[4] Ko´smider,K., Karska,K., Kozakiewicz,A., Lejman,M. and Zawitkowska,J. (2022) Overcoming steroid resistance in pediatric acute lymphoblastic leukemia - the state-of-the-art knowledge and future prospects. Int. J. Mol. Sci., 23, 3795.
[5] Arora, P., Adams, C. H., Gudelsky, G., DasGupta, B. and Desai, P. B. (2019) Plasma and brain pharmacokinetics of letrozole and drug interaction studies with temozolomide in NOD-scid gamma mice and sprague dawley rats. Cancer Chemother. Pharmacol., 83, 81–89.
[6] He, W., Liu, R., Yang, S. H. and Yuan, F. (2015) Chemotherapeutic effect of tamoxifen on temozolomide-resistant gliomas. Anticancer Drugs, 26, 293–300.
[7] Karve, A. S., Desai, J. M., Dave, N., Wise-Draper, T. M., Gudelsky, G. A., Phoenix, T. N., DasGupta, B., Sengupta, S., Plas, D. R. and Desai, P. B. (2022) Potentiation of temozolomide activity against glioblastoma cells by aromatase inhibitor letrozole. Cancer Chemother. Pharmacol., 90, 345–356.
[8] Balça-Silva, J., Matias, D., do Carmo, A., Girão, H., Moura-Neto, V., Sarmento-Ribeiro, A. B. and Lopes, M. C. (2015) Tamoxifen in combination with temozolomide induce a synergistic inhibition of PKC-pan in GBM cell lines. Biochim. Biophys. Acta., 1850, 722–732.
[9] Patel, S., DiBiase, S., Meisenberg, B., Flannery, T., Patel, A., Dhople, A., Cheston, S. and Amin, P. (2012) Phase I clinical trial assessing temozolomide and tamoxifen with concomitant radiotherapy for treatment of high-grade glioma. Int. J. Radiat. Oncol. Biol. Phys., 82, 739–742.
[10] DI Cristofori, A., Carrabba, G., Lanfranchi, G., Menghetti, C., Rampini, P. and Caroli, M. (2013) Continuous tamoxifen and dose-dense temozolomide in recurrent glioblastoma. Anticancer Res., 33, 3383–3389.
[11] Liu, Z., Chen, R., Yang, L., Jiang, J., Ma, S., Chen, L., He, M., Mao, Y., Guo, C., Kong, X., et al. (2024) CDS-DB, an omnibus for patient-derived gene expression signatures induced by cancer treatment. Nucleic acids research, 52(D1), D1163–D1179.




